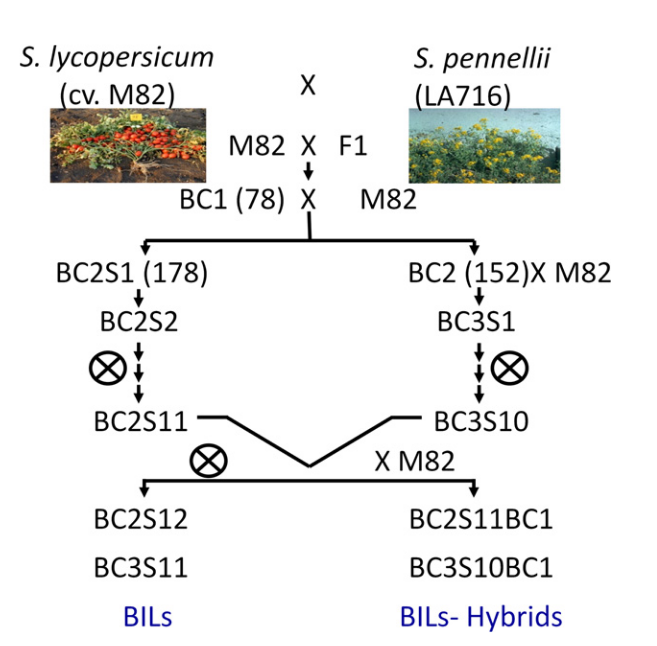
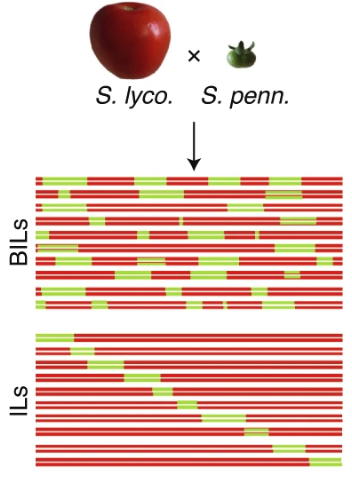
番茄ILs和BILs群体构建
ILs主要是通过LA0716和M82杂交,然后重复回交,最终染色中含有LA0716单一染色体片段。目前,使用的ILs有76个,覆盖了整个番茄基因组,图中A图的黑色线段就是LA0716的染色体片段。这些ILs,为QTL鉴定作出了重要的贡献。
BIL是通过LA0716和M82杂交,然后与M82回交2-3代,最后自己8代,最终得到染色体中有多个LA0716片段,且片段更小,图中为BILs构建的过程。目前,BILs的数量为466个line,相比ILs,能够更精细的确定性状相关区域。
番茄ILs和BILs的主要区别示意图
如何确定目标基因所属的ILs和BILs
换种说法,就是SP基因在M82中表达,有点类似在M82中做了转基因,使其表达SP基因——即寻找包含目标基因的ILs和BILs。
1. 找到目标基因染色体位置
可以在Sol网站中搜索基因,例如基因ID为RIN-Solyc05g012020番茄成熟相关基因。它属于5号染色体,位置是5230962..5225203(-)。

2. 下载BIL-SNP表格
在Solanum pennellii backcross inbred lines (BILs) link small genomic bins with tomato traits文章的supplementary data中下载BIL-SNP表格。表格内容如下:

3. 在BIL-SNP表格中确定包含RIN基因的两个marker
SNP是ch05,位置为5225203-5230962(+)。根据这个信息,在表格中找到包含RIN基因的两个marker,即左侧小于5225203,右侧大于5230962。找到表中位置,两个marker为50687和50718。

4. 根据表格找对应的BILs
我们需要找到表格中数字为3或者0的marker,3表示break point是LA0716的序列,而0表示无信号,可能是可能不是。而1表示M82的序列,2可能是有双峰的意思。

5. 根据ILs染色体上分布图及染色体上的marker,确定包含该基因的ILs
下载文章中https://onlinelibrary.wiley.com/action/downloadSupplement?doi=10.1111%2Ftpj.13194&file=tpj13194-sup-0004-FigS4.TIF (Fig1, FigS4-S14)共12个ILs分布图。
例如RIN是5号染色的ILs分布图,但是这个IL5-1的示意图有些错误
正确的应该是如下图:
我们根据RIN的染色体位置,大概可以判断其是5Mbp左右,考上。marker大概有四个。我们再去搜索BIL-SNP表格表格中搜索这几个marker的位置,看是否搜索到,且包含我们这个RIN基因。
我们发现,snp_997已经是最靠近RIN左边的marker,此时我们还需要向下找marker。
我们首先搜索snp_51230,发现该marker已经可以包括RIN基因。
我们再根据snp-997和snp-51230对应的位置,查看覆盖了那些ILs。结果发现,IL5-2刚好包含了RIN基因,且在5C区域。这样我们就找到了目标ILs。
参考文献
-
Ofner I, Lashbrooke J, Pleban T, Aharoni A, and Zamir D. Solanum pennellii backcross inbred lines (BILs) link small genomic bins with tomato traits. The Plant Journal. 2016:87(2):151–160. https://doi.org/10.1111/tpj.13194
-
Lippman ZB, Semel Y, and Zamir D. An integrated view of quantitative trait variation using tomato interspecific introgression lines. 2007. https://doi.org/10.1016/j.gde.2007.07.007
-
Alseekh S. Resolution by recombination: breaking up Solanum pennellii introgressions. 2013:18(10).